SEP SNEST DGEST
UNIDAD VIII:
REGULACIÓN DE LA EXPRESIÓN GENÉTICA
QUE PRESENTA:
Arisbeth Salgado Mendoza
09930054
Lic. BIOLOGIA VI "A"
Ciudad Altamirano, Gro. México. MAYO del 2012
INTRODUCCIÓN:
Los organismos multicelulares
complejos están compuestos de diferentes tejidos cuyas características
individuales dependen de las proteínas específicas expresadas por sus tipos
celulares. La diferenciación, el desarrollo y la funcionalidad de
los tejidos específicos dependen del conjunto de proteínas selectivamente
expresadas por cada célula. Estas proteínas expresadas en forma diferencial
pueden funcionar como componentes estructurales de las células, enzimas
reguladoras del metabolismo, factores de transcripción, receptores
celulares, componentes intracelulares de señalización, etc.
La expresión incorrecta de tales
proteínas, su expresión en lugares
equivocados, a destiempo,
o la producción en cantidades
anormales de proteínas específicas o de proteínas de función anómala subyace
a toda patología celular de base genética.
Por consiguiente el
conocimiento de los mecanismos de regulación de la expresión proteica en
eucariontes contribuirá al conocimiento de las bases moleculares de
diversas patologías.
La transmisión de la
información genética (transcripción), posibilita la formación de proteínas, cuyas
funciones van a caracterizar la actividad y morfología de las células; pero la
regulación de los tipos y cantidades de proteínas presentes en cada momento
constituye un tema de tanta relevancia como el propio hecho de la síntesis. Las
células son estructuras muy organizadas cuyas moléculas se ordenan de forma
rigurosa.
En un organismo no se
necesitan todos los productos
génicos de forma
simultánea, ni a los mismos
niveles, con lo cual el sistema de regulación va a servir para ajustar el uso
del depósito de información de los ácidos nucleicos a los requerimientos de
cada célula o de cada organismo. A la regulación de la síntesis de las
macromoléculas se la denomina regulación de la expresión genética o génica.
En un organismo procariota,
muy dependiente de las condiciones de su entorno, la regulación debe permitirle
responder rápidamente a las modificaciones medioambientales con el objeto de garantizar
su supervivencia. La utilización de una parte u otra de su dotación genética le
facilitará adaptarse adecuadamente a su entorno. En una célula de Escherichia
coli se
pueden medir niveles diferentes
de concentraciones proteicas; hay proteínas muy escasas, que se encuentran en
número de una decena; y hay proteínas muy abundantes, de las que pueden medirse
miles de copias. En una célula de mamífero pueden existir 1010 moléculas de
proteínas pertenecientes a unos 10.000 a 20.000 tipos diferentes. Esta
enorme variación entre el número y tipo de moléculas presentes es posible por
los cambios controlados en la producción y en la degradación de los productos
génicos.
La diferenciación celular
que existe en los organismos pluricelulares, se fundamenta en que aunque todas
las células disponen del mismo ADN, se distinguen unas de otras porque
sintetizan distintos tipos de moléculas de ARN y de proteínas. Si se compara un
eritrocito y una neurona las diferencias son tan amplias que resulta difícil
pensar que ambas comparten la misma dotación génica. Y si, además, se observa
que la diferenciación es un proceso irreversible, aún se hace más indispensable
el proceso de regulación de la expresión de diferentes genes.
Esta regulación puede
llevarse a cabo en cualquier etapa de la síntesis o de la maduración de las
macromoléculas, y atendiendo a muchas variables se desarrollará en el punto más
adecuado. La vía que conduce desde el ADN hasta las proteínas está formada por
múltiples etapas, y existen pruebas de que todas ellas se pueden controlar. Uno
de los puntos de regulación más importantes se realiza a nivel de la
transcripción, ya que al ser ésta la etapa inicial de trasvase de información
genética se consigue el control en el inicio, lo que supone la mejor pauta de
actuación, ya que se consigue el máximo ahorro energético.
Existen una serie de genes
que se expresan constantemente, denominándoseles genes constitutivos, el
producto de su expresión son moléculas necesarias de forma continua
en todos los momentos de existencia de la célula o del
organismo. Por otro lado, existen otros genes, denominados regulables, cuya
expresión estará ajustada por las necesidades variables de la célula, aumentando
o disminuyendo la expresión génica según se necesite aumentar o disminuir la concentración
del producto génico.
OBJETIVOS:
- Integrará los conocimientos anteriores con los mecanismos de regulación genética para entender a nivel molecular los procesos metabólicos.
METODOLOGÍA:
Con el maestro conoceremos paso a paso el desarrollo de esta unidad la cual presentare en este contesto blooger. definiremos como se da la regulación de la expresion genética a diversos órganos celulares (procariontes y eucariotes).
8.1 NIVELES DE REGULACIÓN DE LA EXPRESIÓN GENÉTICA
Los genes deben ser
regulados ya que no es necesario que se expresen (Transcriban y Traduzcan) todo
el tiempo sino cuando es necesario.
Genes constitutivos y genes
regulados. No todos los genes se expresan simultáneamente ni al mismo nivel.
- Genes constitutivos: los que se expresan al mismo nivel independientemente de las condiciones ambientales
- Genes regulados: aquellos que se expresan a distintos niveles (o no se expresan) dependiendo de las condiciones ambientales.
Los objetivos de la
regulación son:
- Armonía estructural,
equilibrio celular
- Adaptación: típico de
procariotas
- Diferenciación: típico de
eucariotas pluricelulares.
Casi cada uno de los niveles
de los que depende la expresión de información genética puede estar sometido en
principio a algún tipo de regulación, aunque el más frecuente es sobre la
transcripción.
1) Nivel
transcripcional y sobre el ARNm
a) A
nivel del inicio de la transcripción
i) sustitución
del factor s de la ARN-polimerasa
ii) por
interacción de proteínas regulatorias sobre secuencias de ADN cercanas al
promotor:
(1) fenómenos
de inducción génica
(2) fenómenos
de represión génica
A su vez, estos fenómenos
pueden realizarse por:
mecanismos de control
positivo
|
|
mecanismos de control
negativo
|
b) Terminación
prematura de la transcripción: fenómenos de atenuación de la transcripción.
c) Procesamiento
de ARN (casos muy raros en procariotas)
2) Nivel
traduccional. Ejemplos:
a) regulación
de la síntesis de las proteínas ribosómicas.
b) regulación
por ARN antisentido, que interfiere con la traducción del ARNm
3) Nivel
postraduccional. Aquí ya no estamos ante mecanismos puramente de regulación
genética. Algunos ejemplos: degradación de proteínas y modificación covalente
de proteínas (p. ej., fosforilación). Regulación alostérica por
retroalimentación (feed-back) de la actividad de las proteínas enzimáticas.
4) Sistemas
globales de regulación
Cuando varios operones están
regulados coordinadamente por un mismo tipo de estímulos, constituyen una red
de regulación que se suele denominar con el nombre deregulón (p. ej., el
regulón de los operones spo de la esporulación en las especies del
género Bacillus).
Casi todos los productos de
genes bacterianos están regulados en al menos uno de estos niveles, y a menudo
lo están en varios niveles al mismo tiempo.
Como veremos enseguida,
muchos de estos mecanismos se disparan en el interior celular debido a pequeñas
moléculas que informan a la bacteria de un determinado cambio ambiental.
8.2 REGULACIÓN DE LA TRANSCRIPCIÓN EN ORGANISMOS PROCARIOTICOS.
En las bacterias, a pesar de ser organismos unicelulares, también
es necesario regular la expresión de los genes adaptándola a las necesidades
ambientales. Es un principio de economía celular el que la expresión de los
genes este regulada según las circunstancias celulares. Un buen ejemplo de esta
situación en bacterias es la regulación de las enzimas implicadas en el
metabolismo de los azúcares. Las bacterias pueden emplear para obtener energía
distintas fuentes de carbono, como la glucosa, lactosa, galactosa, maltosa,
ramnosa y xilosa. Existen enzimas capaces de introducir cada uno de estos
azúcares en la bacteria y enzimas capaces de romperlos para obtener energía.
Lógicamente, sería un despilfarro energético producir simultáneamente todos los
enzimas necesarios para metabolizar los diferentes azúcares mencionados. Por
consiguiente, sería mucho más económico para la célula producir solamente las
enzimas necesarias en cada momento, es decir, si en el medio en el que vive la
bacteria la principal fuente de carbono es la lactosa, solamente se expresarían
los genes necesarios para metabolizar la lactosa, mientras que los otros genes
no se expresarían. Por tanto, es esencial que exista un mecanismo de regulación
de la expresión génica, de manera que los genes se expresen cuando sea
necesario.
La regulación de la producción de proteínas (síntesis de
proteínas) considerando el proceso en su conjunto, puede llevarse a cabo en
tres niveles:
·
Replicación
·
Transcripción
·
Traducción.
De los tres niveles de regulación, uno de los mejor conocidos
actualmente es la regulación durante la transcripción. Aunque la regulación de
la transcripción en eucariontes es más compleja que en bacterias, muchos de sus
aspectos son similares. Por tanto, comenzaremos por el estudio de la regulación
de la transcripción en bacterias.
8.4.1 OPERON DE LACTOSA (CONTROL POSITIVO)
El operón lactosa es un operón requerido para el transporte y metabolismo de la lactosa en la bacteria Escherichia coli, así como en
algunas otras bacterias entéricas. Presenta tres genes estructurales adyacentes, un promotor, un terminador y un operador. El operón lac es regulado por varios factores,
incluyendo la disponibilidad de glucosa y de lactosa. La regulación génica del operón lac fue el
primer mecanismo regulatorio de la expresión genética en ser elucidado, y es
utilizado a menudo como un ejemplo clásico de la regulación génica en procariotas.
Sistema del operón lactosa.
Un ejemplo claro de regulación de la
expresión genética en procariotas, es el que ocurre en el operón lac. En E. Coli se requieren dos enzimas para la
metabolización de la lactosa, la lactosa permeasa, una enzima de membrana que
permite el transporte de la lactosa al interior celular y la -Galactosidasa,
que cataliza la ruptura del enlace -1,4 entre la galactosa y la glucosa. La lactosa se
degrada mediante rutas catabólicas para la obtención de energía, pero si en el
medio no hay lactosa estas dos enzimas no son necesarias.
La expresión de estas dos enzimas es
inducible por lactosa. ¿Cuál es el mecanismo de esta regulación?
-
El sistema de utilización de lactosa por E.
coli consta de dos clases de componentes (Figura 1). Genes estructurales que
codifican las enzimas -galactosidadas
(gen LacZ), permeasa (gen Lac Y) y
transacetilasa que solo se utiliza en el metabolismo de ciertos -galactosidos
diferetes de la lactosa (gen Lac A), necesarias para el transporte y degradación
de lactosa. Y genes reguladores, el gen Lac I, el promotor lac P y el operador
Lac O y el sito CRP). Menos el Lac I, el resto de genes componen una región del DNA que denominamos operón lac.
Además, los genes estructurales están codificados en un solo mRNA
policistrónico.
El
gen Lac I sintetiza una proteína llamada represor
o inhibidor que se une al operador Lac O de forma que cuando la RNA polimerasa
se una al promotor no pueda continuar la transcripción. De tal forma que el
sistema se encuentra normalmente reprimido y las proteínas necesarias para
metabolizar la lactosa no se sintetizan (Figura 2a). En presencia de
lactosa, un derivado de ésta, la alolactosa, se une al represor cambiándolo
conformacionalmente y haciendo que no tenga afinidad por el operador (la
alolactosa actúa como inductor),
cuando esto ocurre el operador queda libre y la RNA polimerasa puede realizar
la transcripción de los genes. Se trata, por tanto, de una regulación negativa
pues todas las órdenes reguladoras son de prohibición.
No
obstante, se observó además que en presencia de lactosa y glucosa en el medio
de cultivo no había expresión de las proteínas. Esto parece lógico porque si
hay glucosa la bacteria no necesita recurrir a la degradación de lactosa como
fuente de carbono, de tal forma que la glucosa debía regular de algún modo el
proceso. ¿En que consiste la regulación por glucosa? El efecto de la glucosa
está mediado por el AMPc y una proteína llamada proteína receptora de cAMP
(CRP) o proteína activadora de catabolitos.
La
proteína CRP tiene sitios de unión para el ADN y para el AMPc. El complejo
AMPc-CAP se une a una zona cercana al promotor lac, y favorece la unión de la
ARN polimerasa al promotor, aumentando la transcripción hasta 50 veces (Figura
3). Se ha demostrado que el AMPc y la CAP contribuyen a al iniciación de la
transcripción, ayudando al reconocimiento del sitio de iniciación por la ARN
polimerasa.
Los
niveles de AMPc están muy relacionados con la concentración de glucosa, a altas
concentraciones de glucosa los niveles de AMPc son bajos, lo que impide que
éste se una a la proteína CRP, de esta forma la ARN-Pol no puede colocarse en
el promotor y se impide la transcripción de los genes metabolizadores de
lactosa. La función CRP-AMPc favorece la unión de la ARN polimerasa y actúa
como un efector o regulador positivo.
El
AMPc y la CRP están implicados en la regulación de muchos operones,
especialmente los que codifican enzimas para el metabolismo de otros azucáres.
Una red de operones con un regulador común se conoce con el nombre de regulón.
De
forma que se superponen dos regulaciones, la positiva por el efector glucosa y
la negativa por el inductor lactosa que contrarresta el efecto inhibidor del
represor sintetizado por LacI. El operón solo se expresa en presencia de
lactosa y ausencia de glucosa.
Sistema
|
Expresión
|
|
+ Lactosa
|
+ glucosa
|
-
|
+ Lactosa
|
- glucosa
|
+
|
- Lactosa
|
+ glucosa
|
-
|
- Lactosa
|
- glucosa
|
-
|
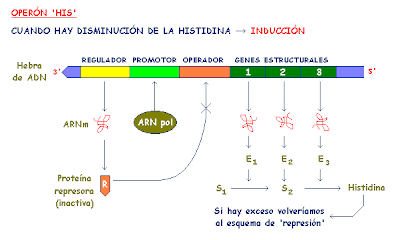
8.4.2 OPERON DE TRIPTOFANO (CONTROL NEGATIVO)
El triptófano es un
aminoácido cuya síntesis se realiza principalmente en diversos operones.
Yanofsky estaba estudiando muy de cerca este operón. Vio que este operón estaba
formado por varios genes, además, observó que cuando había una alta
concentración de Trp no había expresión de los genes, pero que cuado la célula
lo necesita, se expresaban los genes.
Es diferente de la lactosa:
si tenemos Trp no necesitamos más enzimas, y si no lo hay sí.
El represor de este operon
necesita triptófano para poder actuar. El represor es inactivo de por sí, si se
le une Trp se inhibe la transcripción con lo que tenemos un control negativo, y
cuando el efector se une al represor para reprimir es un sistema reprimible.
En este operon hay un gen
que se inducía, el TrpR+. Se aisló un mutante que tenía diez veces más ARNm, y
cuando él fue a mirar a su ARNm descubrió que su mutante tenía una deleción, un
atenuador que cuando estaba atenuaba la expresión.
En el ARNm había una zona
líder que contenía el atenuador, era un ARNm pequeño que estaba en altas
concentraciones. Descubrió que un pequeño péptido correspondiente a esta
secuencia se traducía, mirando su secuencia comprobó que había dos Trp, cosa
que era rara ya que el trp es un aminoácido muy raro. Si había una baja
concentración de triptófano la transcripción continúa. Si los niveles de Trp
eran altos, lee rápido y se une al represor
Los elementos del operón trp
son en esencia semejantes a los del operón lactosa:
• Genes estructurales: existen cinco genes estructurales en el siguiente orden trpE-trpD-trpC-trpB-trpA.
• Elementos de control: promotor (P) y operador (O). El promotor y el operador están al lado de los genes estructurales y en el siguiente orden: P O trpE-trpD-trpC-trpB-trpA. Curiosamente, las enzimas codificadas por estos cinco genes estructurales actúan en la ruta metabólica de síntesis del triptófano en el mismo orden en el que se encuentran los genes en el cromosoma.
• Gen regulador (trpR): codifica para la proteína reguladora. Este gen se encuentra en otra región del cromosoma bacteriano aunque no muy lejos del operón.
•Correpresor: triptófano.
Elementos del Operón Triptófano:
Los genes estructurales del operón triptófano se encuentran en el mismo orden que actúan las productos codificados por ellos en la ruta biosintética del triptófano
Orden de los genes estructurales del operón triptófano y ruta de síntesis del triptófano
En ausencia de triptófano, o cuando hay muy poco, la proteína reguladora producto del gen trpR no es capaz de unirse al operador de forma que la ARN-polimerasa puede unirse a la región promtora y se transcriben los genes del operón triptófano.
8.3 REGULACIÓN DE LA TRANSCRIPCIÓN EN ORGANISMOS EUCARIOTES
En el caso de las eucariotas, el proceso se realiza en el núcleo, y es similar al
de las procariotas, pero de mayor complejidad. Diferentes ARNp transcriben
distintos tipos de genes. La ARNpII transcribe los pre-ARNm, mientras que la
ARNpI y ARNpIII transcriben los ARN-ribosomales y ARNt, respectivamente. Los
ARNs transcritos son modificados posteriormente. El pre-ARNm,por ejemplo, sufre
un proceso de maduración que tras cortes y empalmes sucesivos elimina ciertos
segmentos del ADN llamados los intrones para
producir el ARNm final. Durante este proceso de maduración se puede dar lugar a
diferentes moléculas de ARN, en función de diversos reguladores. Así pues, un
mismo gen o secuencia de ADN, puede dar lugar a diferentes moléculas de ARNm y
por tanto, producir diferentes proteínas. Otro factor de regulación propio de
las células eucariotas son los conocidos potenciadores (en inglés: "enhancers"), que
incrementan mucho (100 veces) la actividad de transcripción de un gen, y no
depende de la ubicación de éstos en el gen, ni la dirección de la lectura.
Activadores
transcripcionales
Los
activadores son la proteínas que se van a unir a los elementos distales (SDE y
potenciadores) para activar la transcripción. Son específicos de unos pocos promotores —por lo que no estarán en
todos los tipos celulares—, reconocen entre 6 y 14 pb en el promotor y suelen tener dos dominios estructurales:
- El
dominio de unión a DNA (DNA
binding domain) , que consta de 60 a 100 aminoácidos consecutivos.
- El
dominio de activación de la
transcripción que consta de 30 a 100 aminoácidos que no tienen por qué ser
consecutivos.
- La
presencia de estos dominios las convierte en proteínas modulares en las
que el dominio de unión y el de activación pueden funcionar
independientemente.
2.-
Coactivadores y correpresores
coactivador si ayuda a activar la transcripción.
Un mismo coactivador puede recibir señales de distintos activadores para
transmitirlos hacia el complejo del promotor basal.
correpresor si ayuda a inactivar el promotor. Los
correpresores pueden tener actividad desacetilasa, con lo que hace que el DNA
se una con más firmeza a los nucleosomas, inactivando el promotor porque no
puede ser reconocido por los factores generales de transcripción.
3.- Transactivadores
Son
aquellos que directamente ejercen su acción interaccionando con el complejo de
iniciación formado en el promtor basal, bien sobre la propia polimerasa o, más
normalmente, sobre una de las TAF o de los TFII, para activar o reprimir la
transcripción, puesto que no son actividades exclulyentes.
Potenciadores
los potenciadores (enhancers
o intensificadores). Su función es la de amplificar la transcripción del
promtor incluso más de 1000 veces. Los hay específicos del tejido —sólo activan la transcripción de su gen en
determinados tejidos—, específicos de la
etapa de desarrollo
e inducibles por alguna señal externa como hormonas, metales pesados, choque
térmico, infección viral, etc. Necesitan la mediación de un coactivador.
Silenciamiento de genes
La
unión inespecífica de proteínas reguladoras es un problema importante en los
organismos con genomas grandes. Para combatirla, los eucariotas han hecho que
los genes tengan en torno a 5 dianas para proteínas reguladoras diferentes.
Esta estrategia es útil para los activadores de la transcripción porque es una
estrategia eficiente y ahorra esfuerzo. Una estrategia similar no es factible
con los inhibidores de la transcripción, por lo que se da poca regulación por
silenciamiento.
El silenciamiento de un gen puede
ocurrir por:
- la
inactivación por interacción con un regulador
- el
silenciamiento génico postranscripcional (PTGS, también denominada
cosupresión o extinción génica)
- la
metilación del DNA en vertebrados (directamente ligada al
superenrollamiento y al silenciamiento).
Inactivación
mediante una proteína reguladora
Se
consigue uniendo una proteína reguladora a cualquiera de los distintos
elementos que forman los promotores.
Los
que reconocen
los elementos distales
• El silenciador específico
de tejido (tse): se encarga de silenciar en cualquier célula los genes
que son específicos de células hepáticas
• Las hormonas esteroideas comentadas anteriormente
• El gen Pit-1
Silenciamiento
por metilación
No
todos los organismos tienen el DNA metilado. En los mamíferos, el DNA metilado
forma heterocromatina a la que no pueden acceder los factores de transcripción.
Por tanto, los genes metilados no se pueden transcribir ni tan siquiera
residualmente. Se trata de un mecanismo muy eficiente de silenciamiento génico
que, además, disminuye la cantidad de DNA que los factores de transcripción y
la RNA-polimerasa tienen que rastrear para buscar los promotores.
TAREA: Investigue como se realiza el control genético del "Gen BRCA".
Es un gen humano del tipo del gen supresor de tumores, que regulan el ciclo celular y evitan la
proliferación incontrolada.
La proteína BRCA1, producto de este gen, forma parte del sistema de
detección y reparación de los daños del ADN. Las variaciones de este gen están implicadas en
algunos tipos de cáncer, especialmente el cáncer de mama. El gen BRCA1 está
situado en el brazo largo (q) del cromosoma 17, en la posición 21, desde el par de bases
38.449.843 hasta el par de bases 38.530.933.
El concepto de
la realización de pruebas genéticas para el cáncer de mama procede del
reconocimiento de que existen cúmulos de casos de cáncer de mama en familias, a
veces asociados a cáncer de ovario en las mujeres o cáncer de próstata en los
varones.
No fue hasta
principios de la década de 1990 cuando se describieron dos genes de cáncer de
mama de elevada penetrancia. Los oradores estimaron que estos genes, BRCA 1 y 2,
explican el mayor riesgo de cáncer de mama y ovario en el 20% al 50% de las familias.
Las mujeres portadoras de una estas mutaciones poseen un riesgo de cáncer de
mama a lo largo de la vida de hasta el 50% al 80%, y un riesgo de entre el 15%
y el 25% de cáncer de ovario. También podría asociarse a estas mutaciones
genéticas un riesgo elevado de cáncer de próstata y quizá de otros cánceres,
incluyendo el de páncreas. Desde principios de la década de 1990, cuando
se describieron los genes BRCA 1 y 2, no se han identificado nuevos genes
de este tipo.
De acuerdo con
el Dr. Stratton, sólo se puede atribuir a BRCA 1 y 2 el 20% del riesgo
familiar de cáncer de mama. Los genes asociados al cáncer de mama se pueden
dividir en uno de los tres tipos siguientes: de riesgo elevado, que comportan
un riesgo de 20 a lo largo de la vida; de riesgo moderado, que comportan un
riesgo de 5-10 a lo largo de la vida, y los de bajo riesgo. Para
identificar los genes de alto riesgo, se puede emplear el análisis de
ligamiento genético en un pequeño número de grandes familias con muchos
miembros afectados. Es este tipo de estudio el que condujo a la identificación
de los genes BRCA 1 y 2. El gen BRCA 2 se encontró y localizó sin ambigüedades
en una única familia de gran tamaño. Se ha propuesto que existen loci de
BRCA 3 de riesgo alto a moderado en el cromosoma 8p y 13q21. El Dr. Stratton
afirmó que cree que no existe evidencia de un gen de vulnerabilidad de alto
riesgo en ninguno de estos dos loci.
http://www.cirugest.com/htm/revisiones/cir09-06/09-06-20.htm
CONCLUSIONES:
con el profesor vimos paso a paso detenidamente los puntos a conocer de como se daba la regulación de la expresión genética en los diferentes tipo de organismos (EUCARIOTICOS y PROCARIOTICOS), en ellos estudiados los operones encontrados en dichas partes.
BIBLIOGRAFIA:
- http://www.ucm.es/info/genetica/grupod/Operon/Operon.htm#Regulación en bacterias
- http://www.ugr.es/~eianez/Microbiologia/15regulacion.htm
- http://www.google.com.mx/url?sa=t&rct=j&q=&esrc=s&source=web&cd=6&ved=0CGEQFjAF&url=http%3A%2F%2Fwww.unioviedo.es%2Fbos%2FAsignaturas%2FFvca%2Fseminarios%2FRegulacindelaexpresingnica.doc&ei=7ta-T5S9FoPQ2wWs4vz_CQ&usg=AFQjCNFwsAR9L-Z7OAnxwJ695Gc8XxXR0Q&sig2=QC1CEdqghFsMKdPyjmL9zQ
- http://ocw.unican.es/ciencias-de-la-salud/fisiologia-general/materiales-de-clase-1/tema-1.-introduccion-al-estudio-de-la-fisiologia/Tema%208-Bloque%20I-Regulacion%20Genetica.pdf
- http://www.ramc.cat/composicio/Santiago%20Vidal%20Sivilla.pdf
- http://www.cienciaybiologia.com/bgeneral/regulacion-expresion-genica-procariotas-virus.htm
- http://genmolecular.wordpress.com/regulacion-de-la-expresion-genica/