SEP SNEST DGEST
UNIDAD IV:
REPLICACION DEL ADN
QUE PRESENTA:
Arisbeth Salgado Mendoza
09930054
Lic. BIOLOGIA VI "A"
Ciudad Altamirano, Gro. México. MARZO del 2012
INTRODUCCIÓN:
Es el
mecanismo que permite al ADN duplicarse
(es decir, sintetizar una copia idéntica). De esta manera de una molécula de
ADN única, se obtienen dos o más "clones" de la primera. Esta
duplicación del material genético se produce de acuerdo con un mecanismo semiconservativo, lo que indica que las dos cadenas
complementarias del ADN original, al
separarse, sirven de molde cada una para la síntesis de una nueva cadena
complementaria de la cadena molde, de forma que cada nueva doble hélice contiene una de las cadenas del ADN original.
Gracias a la complementariedad entre las bases que forman la secuencia de cada una de
las cadenas, el ADN tiene la
importante propiedad de reproducirse idénticamente, lo que permite que la
información genética se transmita de una célula madre a
las células hijas y
es la base de la herencia del material genético.
La molécula de ADN se abre como
una cremallera por ruptura de los puentes de hidrógeno entre las bases complementarias liberándose dos hebras
y la ADN polimerasa sintetiza la mitad complementaria
añadiendo nucleótidos que se encuentran dispersos en el núcleo. De esta forma, cada nueva molécula es
idéntica a la molécula de ADN inicial.
La replicación empieza en puntos determinados: los orígenes de replicación.
Las proteínas iniciadoras reconocen secuencias de nucleótidos específicas en esos puntos y
facilitan la fijación de otras proteínas que permitirán la separación de las
dos hebras de ADN formándose
una horquilla de replicación. Un gran número de enzimas y proteínas intervienen en el mecanismo molecular
de la replicación, formando el llamado complejo de replicación o replisoma. Estas proteínas y enzimas son
homólogas en eucariotas y arqueas, pero difieren en bacterias
OBJETIVOS:
- Entenderá como se
transmite la información genética entre los seres vivos y su relación con los
mecanismos de herencia.
- Distinguirá los
mecanismos de duplicación del material genético en procariotas y eucariotas.
METODOLOGÍA:
Se llevara acabo la realización por medio de como vamos viendo la unidad lo aprendido y lo investigado de la unidad ya anexando tareas y trabajos en el, requerido por el profesor.
TAREA:
El experimento de Meselson-Stahl fue
un experimento realizado en 1957 por Matthew Meselson y Franklin Stahl en el que se demostró que la replicación de ADN era semiconservadora.
Una replicación semiconservadora es aquella en que la cadena de dos filamentos
en hélice del ADN se replica de forma tal que cada una de las dos cadenas de ADN formadas
consiste en un filamento proveniente de la hélice original y un filamento nuevo
sintetizado.
El
experimento permitió confirmar las teorías de James Watson y de Francis Crick sobre el método de replicación del ADN.
Las figuras
siguientes ilustran el experimento Meselson y Stahl que demostró lo correcto
del modelo semiconservativo de la replicación del ADN. La centrifugación del
CsCl (cloruro de cesio) y la banda o
bandas del ADN para cada condición de cultivo, comparado con la banda control,
marcada como 14N (ligera) del ADN.
- Las bacterias crecen en el
medio 14N o 15N.
- Muestras removidas y el ADN
extraído.
- Centrifugue las muestras de
ADN.
Descripción del experimento Meselson y Stahl
Meselson y Stahl probaron la hipótesis de la replicación del ADN. Ellos
cultivaron bacterias en un medio 15N. El 15N es un isótopo pesado de nitrógeno,
por lo tanto el ADN sintetizado es de densidad pesada. Ellos entonces cambiaron
las bacterias al medio 14N. El ADN se aisló diferentes veces que corresponden a
los ciclos de replicación 0, 1, y 2. Después de un ciclo de replicación, el ADN
fue todo de densidad intermedia. Esto descarta el modelo conservador de la
replicación, que predice que ambos ADN (pesado y liviano) estarán presentes,
pero ninguno de densidad intermedia estará presente.
Este resultado es consistente con el modelo semiconservativo de replicación,
que predice que todas las moléculas de ADN consistirán de una cadena 15N de ADN
y una cadena 14N de ADN.
El resultado no descarta el modelo dispersivo de replicación, que también
predice que todo el ADN será de densidad intermedia, consistiendo de segmentos
intercalados de doble cadena 15N y 14N.
Después de dos ciclos de replicación, se ven dos bandas de ADN, una de densidad
intermedia y una de densidad liviana. Este resultado es exactamente lo que el
modelo semiconservativo predice: la mitad debería ser densidad intermedia
15N-14N la intermedia y la mitad de densidad liviana 14N-14N.
Este resultado descarta el modelo dispersivo de la replicación, que predice que
después del ciclo 1 de replicación, la densidad del todas las moléculas de ADN,
gradualmente llegarían a ser más bajas. Por lo tanto, ningún ADN de densidad
intermedia intermedio debería permanecer después del ciclo 2. El modelo
semiconservativo es correcto.
http://www.maph49.galeon.com/adn/experiment.html
http://es.wikipedia.org/wiki/Experimento_de_Meselson-Stahl
4.1 REPLICACION DEL GENOMA PROCARIOTICO.
Es
un mecanismo presente en organismos de genoma reducido, tales como
bacteriófagos. Son genomas que pueden necesitar en un momento dado muchas
replicaciones. Un ejemplo de este mecanismo, lo sufre el fago X174,
produciéndose sus concatémeros mediante
este tipo de replicación; suele servir para replicar pequeños genomas decadena sencilla circulares, tales
como fragmentos extracromosómicos portadores de agrupaciones de genes para elrRNA y
para factores F de
plásmidos bacterianos. La replicación de este genoma, la podemos dividir en
tres fases principales.
Cuando el virus infecta la célula huésped, el
genoma se introduce en la bacteria y lo transforma a cadena de DNA
monocatenaria, que recibe el nombre de cadena +, se convierte en una doble hélice o forma replicativa RFdespués de la
síntesis de la cadena de DNA complementaria, que recibe el nombre de cadena -, gracias a DNA polimerasa
III y los cebadores.
La forma replicativa bicatenaria se replica varias
veces y produce poblaciones de moléculas
RF.
Por último, estas poblaciones se replican
asimétricamente y dan lugar a una progenie de moléculas monocatenarias+. Estas últimas se
asocian con las proteínas de cubierta del fago y completan su ciclo
reproductivo. En las dos últimas fases, la síntesis de DNA tiene lugar por el
mecanismo de círculo rodante.
El
proceso del círculo rodante se inicia con una actividad endonucleasa específica de secuencia que permite a la
denominada proteína A romper
la cadena positiva de la forma replicativa
parental RF en un punto que puede considerarse como origen de la
replicación. Su acción provoca extremos 3´-OH y 5´ fosfato
en esta cadena, mientras que la cadena - queda
intacta. El extremo libre 5´ fosfato
va siendo desenrollado y liberado de la molécula, a la vez que la cadena - gira sobre su
eje y forma una especie de círculo con una pequeña cola. A mesura que el
círculo va girando sobre sí mismo, el extremo 5´ va siendo desplazado, liberado
de la molécula, mientras la polimerasa va elongando lacadena + desde el extremo
3´-OH, usando como molde la cadena
-. Como resultado de uno de estos ciclos, obtenemos una nueva forma
replicativa parental abierta en la cadena +, junto con un DNA monocatenario de tipo cadena +. Estacadena + es el genoma del
virus, de forma que esta puede volver a convertirse en RF. Esta cadena + puede volver a
dar otra (-)
discontinuamente, gracias a la DNA
polimerasa III y I.
Esta
hipótesis surgió debido a que la organización del genoma mitocondrial es
radicalmente diferente del genoma nuclear. Los genomas mitocondriales presentan
varias características de los genomas procariotas como:
§ Pequeño
tamaño.
§ Ausencia
de intrones.
§ Porcentaje
muy elevado de ADN codificante.
§ Falta
generalizada de secuencias repetidas y genes de rARN comparativamente pequeños,
parecidos a los de procariotas.
La
evolución del código genético mitocondrial es probablemente el resultado de una
presión de selección reducida en respuesta a una capacidad codificante muy
disminuida.
En
bacterias, la división celular ocurre justo tras la replicación del cromosoma,
incluso muchas veces, antes de terminar la replicación comienza la división,
pero en los eucariotas, tenemos entre estos dos pasos, la fase G2. Esto ocurre incluso en células
con gran desarrollo, de forma que todas estas dificultades vitales hacen que
los eucariotas se enfrenten a problemas que no poseen las bacterias.
Una
de ellas es como se compatibiliza la actuación de 2 DNA polimerasas en la replicación
de eucariota. También como segregan los cromosomas en las células hijas, de
forma que otro aspecto es como se resuelve la replicación de zonas lineales de
los cromosomas.
Encontramos
enzimas similares en eucariota y procariota. El origen de replicación se SV40 es una región de 64
pares de bases, con secuencias concretas y particulares. Tenemos un antígeno T (dos hexámeros de antígeno T), que reconoce la
secuencia Ori e
interacciona con ella, de forma que encontramos actividad helicasa en el antígeno T, la
cual para ser activada necesita la proteína RF-A que se encuentra en el huésped. Una vez ocurre la
activación con gasto energético de ATP, también actuaría como proteína de unión
a las cadenas sencillas SSB,
de forma que a diferencia de procariota, el inicio de replicación es en la
región palindrómica rica en A=T,
también.
4.2 REPLICACION DEL GENOMA EUCARIOTICO
El término genoma se usa para referirse a todos los alelos que posee un
organismo (o una población, especie, o un gran grupo taxonómico). Si bién la
cantidad de ADN de una célula diploide es constante para una especie, existen
grandes diferencias entre la mismas. Homo sapiens tiene 3.5 X 109 pares de
bases y la Drosophila tiene 1.5 X 108, por genoma haploide. Mucho del ADN de
cada célula no tiene función o bién la misma no es conocida. Solo el 10% del
ADN eucariota codifica para proteína . Los virus y procariotas aparentemente
usan mucho mas sus ADN.
Practicamente la mitad del ADN eucariota
corresponde a secuencia nucleotídicas repetidas. Las secuencias que codifican
proteínas estan interrumpidas por regiones que no codifican. Las secuencias no
codificantes se denominan intrones. Las codificantes exones.
Los nucleótidos trifosfatos (en la formas de ATP, GTP,
CTP y TTP) se enganchan de acuerdo al modelo semi-conservativo . Otros detalles
de la replicación del ADN son consistentes con los hallazgos en procariotas. El
ADN eucariota tiene muchas horquillas de replicación y también síntesis
bidireccional. El ADN eucariota se sintetiza mucho mas lentamente que el
procariota, en humanos, a unos 50 nucleótidos por segundo y por horquilla de
replicación. Luego de la replicación el nuevo ADN se asocia inmediatamente con
histonas.Para una detallada descripción del proceso consultar el Curtis H.
(Editorial Panamericana, 5ta. edición), pag. 317, fig.14-18 y fig. 14-19.

El
mecanismo es similar al que acabamos de ver en procariota. Es una replicación
semiconservativa y bidireccional, de forma que también necesitamos un cebador.
Una vez se inicia la replicación de un replicón (tramo de DNA que se replica a partir de un
mismo origen de replicación), de forma que una vez se inicia la replicación
continúa hasta el final. En E.Coli sólo
existe un replicón, pero conforme avanzamos en organismos eucariotas, podemos
encontrar un mayor número de replicones, observando que son bastante más
pequeños que los de E.Coli. Mientras que en levadura tenemos unos
500, un ratón puede llegar a tener 25000. No todos los replicones de una misma
célula funcionan al mismo tiempo, sino que a lo largo de la replicación existe
una ordenación temporal en cuanto al momento en que se replican. El que
organismos superiores posean tal cantidad de replicones se debe a la gran
cantidad de material hereditario de estos organismos, de forma que son
utilizados para que el proceso no se alargue excesivamente.
Encontramos
en eucariota una gran cantidad de inicios
de replicación distribuidos a lo largo del genoma. En cada inicio
se produce la desnaturalización del DNA y su replicación bidireccional. La
replicación se lleva a cabo a partir de 2DNA polimerasas que trabajan sincrónicamente en la horquilla
de replicación. En levadura se
han observado secuencias de 200 pares de bases que pueden ser consideradas los
inicios de replicación, pues además coinciden en número con el número de
replicones . Además, cuando las añadimos a moléculas circulares de DNA
extracromosómico, confieren a esta capacidad para auto-replicarse de forma autónoma dentro de la célula de
levadura. En algunos animales también se han identificado secuencias de este
tipo. Estas secuencias se denominan secuencias de replicación autónomas o ARS. Son secuencias ricas en pares A=T, dando inestabilidad que
provoca la apertura de las zonas.
En
eucariota, podemos encontrar 5 polimerasas distintas, que son , , , , . Las
polimerasas , , han
sido descritas en el núcleo, mientras que la polimerasa parece ser
mitocondrial. , replican
el DNA nuclear (equivalentes a DNA polimerasa I y III), mientras que la interviene
en reparación.
La
polimerasa posee cuatro subunidades, una de las cuales inhibe la
actividad exo 3´!5´,
de forma que si no se separa no tendrá la capacidad de corrección de errores.
Otras dos subunidades presentan actividad
primasa, factor importante que diferencia la replicación eucariota
de la procariota, en este caso, la actividad primasa no es independiente de la
polimerasa. La cuarta subunidad es la DNA polimerasa propiamente dicha. Como en
todos los casos, la actividad exonucleasa 3´!5´ y la 5´!3´ se encuentran en el amino terminal, mientras que la
actividad polimerásica se encuentra en el extremo carboxilo terminal. La
polimerasa sólo está formada por dos subunidades, y en presencia de
una proteína auxiliar denominada ciclina o PCNA, es mucho más activa y
procesiva que la polimerasa . Se ha propuesto un mecanismo de del DNA
eucariota, según el cual la polimerasa unida a PCNA sintetizaría
la cadena adelantada, mientras que la polimerasa , junto con la primasa,
sintetizaría los fragmentos de okazaki de la cadena retrasada, los cuales son
más pequeños que los procariotas.
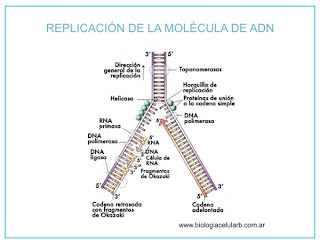
4.3 CONTROL GENÉTICO DE LA REPLICACION
4.4 REPLICACION IN VITRO DEL ADN (PGR)
Para que una
especie no se extinga los individuos deben reproducirse, con el fin de
engendrar nuevos seres. De la misma manera, para que una célula pueda dividirse
es necesario que primero duplique su material genético y así poder garantizar
la misma dotación cromosómica a las células hijas. El modelo de la doble hélice
de Watson y Crick permitió explicar cómo las moléculas de ADN pueden copiarse,
es decir, replicarse y dar una molécula idéntica al molde o patrón.
Hipótesis de la
duplicación del ADN
·
Hipótesis
semiconservativa: formulada por Watson y Crick. En una doble hélice
cada hebra servirá de molde y, mediante la complementariedad de bases, se
formará una hebra copia de cada hebra molde, quedando al final dos dobles
hélices formadas por una hebra antigua (molde) y una hebra nueva (copia). En
1957, experimentos realizados por Meselson y Stahl confirmaron esta
hipótesis.
·
Hipótesis conservativa:
tras la duplicación quedan dos hebras antiguas y dos hebras nuevas formando una
doble hélice.
·
Hipótesis dispersa:
se propone que las hebras están formadas por fragmentos distintos de ADN
antiguo y ADN recién sintetizado.
El crecimiento de las
nuevas hebras
·
La ADN-polimerasa:
El estudio in vitro de la
duplicación del ADN fue posible gracias al aislamiento de la enzima ADN-polimerasa porKornberg. Esta enzima es incapaz
de iniciar una cadena de novo; requiere la
presencia de un extremo libre del carbono 3' de un nucleótido, para poder ir
añadiendo los nucleótidos nuevos. Este extremo 3' libre lo aporta el cebador o
«primer», que es una porción pequeña de nucleótidos complementaria al extremo
de la cadena patrón. Por tanto, la cadena naciente siempre crecerá en el
sentido 5'®3'. El primer nucleótido de la cadena nueva tiene un
extremo 5' libre; se irán añadiendo nucleótidos a los extremos 3' libre y se
irán formando los enlaces fosfodiester 3'®5', de forma que el
último nucleótido tendrá libre el carbono 3'.
Los nucleótidos se añadirán siguiendo las reglas de
la complementariedad de bases, de manera que la nueva hebra sintetizada será
antiparalela y complementaria a la patrón.
·
La duplicación del ADN in
vivo:
Estudios realizados con bacterias comprobaron que
el cromosoma bacteriano tenía un origen
de replicación, un punto en el ADN circular donde se iniciaba la
síntesis de las hebras nuevas. Este punto se encontraba en una burbuja de replicación, donde se
abría la doble hélice, y formaba lo que se denominaron las horquillas de replicación.
En el mecanismo de duplicación in vivo surgían dos dilemas: ¿cómo podía la
ADN-polimerasa iniciar la polimerización sin cebador? Y si la ADN-polimerasa
sólo añadía nucleótidos en la dirección 5'®3', ¿cómo se explicaba
el crecimiento, en sentido 3'®5', de una de las
hebras de la horquilla de replicación?
La solución la dio Okazaki:
encontró fragmentos de mil a dos mil nucleótidos de ADN y unos cincuenta
nucleótidos de ARN, que se añadían discontinuamente sobre la hebra patrón. A
medida que se abría la horquilla de replicación se iniciaba la síntesis de un
nuevo fragmento de Okazaki. Una hebra
de la horquilla se copiaba de forma continua en dirección 5'®3' y la otra lo hacía de forma discontinua, mediante los
fragmentos de Okazaki, también dirección 5'®3'. Los nucleótidos de
ARN eran añadidos por la ARNpolimerasa, enzima que no precisa cebador, y luego
la ADN-polimerasa iba incorporando los desoxinucleótidos, sobre la hebra
patrón.
Mecanismos de duplicación
del ADN
Aunque existen algunas diferencias el proceso es
básicamente igual en bacterias y en eucariotas:
·
La secuencia de nucleótidos en el origen de
replicación del ADN actúa como señal
de iniciación.
·
La enzima helicasa separa
las dos hebras de la doble hélice para que sirvan de molde. El desenrollamiento
de la hélice da lugar al superenrollamiento en los extremos de la horquilla de
replicación, actuando entonces las enzimastopoisomerasas que
liberan esta tensión. La topoisomerasa I corta una hebra y la topoisomerasa II
(denominada girasaen E. coli) las dos. Una vez liberada la tensión vuelven a
sellar la doble hélice.
·
Mientras se separan las dos hebras se van uniendo
las proteínas estabilizadoras (SSB),
de forma que se mantengan separadas ambas hebras y se estabilice la horquilla
de replicación.
·
El proceso de duplicación es bidireccional; hay dos horquillas
de replicación por cada burbuja de replicación.
·
La primasa (una
ARN-polimerasa) sintetiza los fragmentos de ARN que sirven de cebador (primer) para la ADN-polimerasa.
·
La ADN-polimerasa III incorpora en dirección 5'®3' los nucleótidos, formando una nueva hebra de crecimiento continuodenominada
hebra conductora.
·
Sobre la otra hebra antiparalela, primero, a unos
mil nucleótidos del origen de replicación, se sintetizarán unos cincuenta
nucleótidos de ARN que servirán para que la ADN-polimerasa III incorpore los
desoxinucleótidos, formándose los fragmentos de Okazaki a medida que se va
abriendo la horquilla. Una vez formados, la ADN-polimerasa
I, gracias a su función exonucleasa, irá eliminando los tramos de ARN y
los irá rellenando con ADN, sintetizados gracias a su actividad polimerasa.
·
Finalmente interviene la ADN-ligasa, que empalma entre sí
los distintos fragmentos de la hebra de crecimiento discontinuo, denominada hebra retardada.
4.4.1 SECUENCIACION DEL ADN
Las
posibles letras son A, C, G, y T,
que simbolizan las cuatro subunidades de nucleótidos de una banda ADN - adenina, citosina,guanina, timina, que son bases covalentemente
ligadas a cadenas fosfóricas. En el típico caso, las secuencias se presentan
pegadas unas a las otras, sin espacios, como en la secuencia AAAGTCTGAC, yendo
de 5' a 3' de izq. a derecha.
Una
sucesión de cualquier número de nucleótidos mayor a cuatro es pasible de
llamarse una secuencia. En relación a su función biológica, que puede depender
del contexto, una secuencia puede tener sentido o antisentido,
y ser tanto codificante o no codificante. Las
secuencias de ADN pueden contener "ADN no
codificante."
Las
secuencias pueden derivarse de material biológico de descarte a través del
proceso de secuenciación de ADN.
En
algunos casos especiales, las letras seguidas de A, T, C, y G se presentan en
una secuencia. Esas letras representan ambiguedad. De todas las moléculas
muestreadas, hay más de una clase de nucleótidos en esa posición. Las reglas de
la Unión Internacional de Química Pura y Aplicada (IUPAC)
son las que siguen:
A
= adenina
C
= citosina
G
= guanina
T
= timina
R
= G A (purina)
Y
= T C (pirimidina)
K
= G T (keto)
M
= A C (amino)
S = G C (enlaces fuertes)
W = A T (enlaces débiles)
B
= G T C (todos y A)
D
= G A T (todos y C)
H
= A C T (todos y G)
V
= G C A (todos y T)
N
= A G C T (cualquiera)
Las secuencias de ADN son una especie de texto de instrucciones, ya que
del orden de las letras A, C, G, T depende el orden de otros polímeros en
nuestras células. Es decir, la secuencia de ADN es interpretada por la célula
para la construcción de otros polímeros, los cuales son cruciales para las
funciones celulares. En concreto, la secuencia de ADN, a través de una molécula
intermediaria relacionada en composición llamada ARN (Ácido RiboNucléico),
determina la secuencia de las llamadas proteínas. Los monómeros que forman a
las proteínas son llamados aminoácidos, pero los aminoácidos son 5 veces más
numerosos que los nucleótidos; es decir, hay 20 aminoácidos
diferentes. Las secuencias de ADN (mediante el ARN) definen cuales de los 20
aminoácidos se usarán para la proteína y también cuál será su orden o
secuencia. La composición y orden de los aminoácidos en las proteínas es
fundamental para sus propias características, de manera que la secuencia de
aminoácidos de la proteína Mioglobina, que transporta oxígeno en nuestros
músculos, es muy distinta a la secuencia de aminoácidos de la proteína
Rodopsina en nuestros ojos, que nos sirve para detectar la luz. Aunque, hay
también no pocos casos en los cuales proteínas de secuencia de aminoácidos
distintas tienen funciones muy semejantes.
En la actualidad es de mucha importancia saber los detalles de las secuencias
de ADN y del proceso de formación de proteínas al cual, dicho sea de paso, le
llamamos traducción (con ARN como intermediario), como para los lenguajes, y en
éste sentido la analogía es muy válida ya que las “letras” que forman al ADN (y
al ARN) son distintas a las “letras” que forman a las proteínas, así que sería
como traducir de un lenguaje a otro.

CONCLUCION:
Llegamos a la conclusion de que conocimos por medio de la practica y lo que nos explico el profesor como podemos dividir nuestro propio ADN y ver de que procedimiento esto se divide en su secuencia.
BIBLIOGRAFIA:
- http://es.wikipedia.org/wiki/Replicaci%C3%B3n_de_ADN
- http://html.rincondelvago.com/replicacion-del-adn_1.html
- http://minnie.uab.es/~veteri/masters/Tema%202%20-%20Genoma-proc.pdf
- http://www.hiru.com/biologia/la-duplicacion-del-adn
- http://hypatia.morelos.gob.mx/index.php?Itemid=419&id=489&option=com_content&task=view

























